Articles destacats

Las tácticas más prometedoras para vencer la guerra contra las bacterias

Iniciativa Resistencia a Antibióticos de Isglobal*
última actualización: 11 de noviembre de 2016
Mucho se ha dicho y escrito en estos últimos años sobre el aumento alarmante, a nivel global, en el número de bacterias de relevancia médica que son resistentes a uno o varios antibióticos debido al abuso y mal uso de antibióticos no sólo en humanos, sino también en el sector agropecuario: en los últimos 30 años el uso de antibióticos en animales de granja ha aumentado entre 600% (antibióticos relacionados a la penicilina) y 1.500% (tetraciclinas).
El problema de la resistencia antimicrobiana es, en cierta manera, comparable al del cambio climático: es resultado de la conducta poco responsable del hombre y puede tener consecuencias drásticas para la humanidad. Sin embargo, como con el cambio climático, tenemos el conocimiento y herramientas para poder hacer frente al problema y ganarle la guerra a la resistencia antimicrobiana.
En este informe digital queremos resaltar lo que se está haciendo y logrando en cuatro frentes fundamentales en la batalla contra las bacterias resistentes: la búsqueda de nuevos antibióticos, los avances en el diagnóstico microbiológico, el estudio de los mecanismos de resistencia, y la vigilancia de propagación y emergencia de resistencias microbianas.
1. Nuevas armas
(nuevos antibióticos)
¿Por qué es un campo de batalla fundamental?
Para hacer frente al problema de resistencia antibacteriana, necesitamos nuevos antibióticos y los necesitamos rápidamente. Y sin embargo, el desarrollo de nuevos antibióticos por las grandes compañías farmacéuticas está prácticamente parado. Desde 1987, no se ha descubierto ninguna clase nueva de antibióticos y de los 8 antibióticos aprobados por la agencia estadounidense Food and Drug Administration (FDA) en los últimos 6 años, la mayoría no tienen nuevos mecanismos de acción, o son simplemente combinaciones de antibióticos ya conocidos. Es como si en pleno siglo XXI usáramos bayonetas para combatir a un ejército equipado con cohetes teledirigidos.
¿Qué tácticas se están empleando?
a) Adaptar las armas que ya tenemos
Una táctica para tratar infecciones por bacterias resistentes a antibióticos es la recuperación de antibióticos “olvidados”, como la colistina, para sacarles mayor provecho y “aliviar” el uso de antibióticos más recientes, o bien la “alternancia” de antibióticos en hospitales. Sin embargo, esto no es una solución a largo plazo ya que la aparición de cepas resistentes es inevitable.
Otra táctica que se está llevando a cabo es la de combinar fármacos que tienen diferentes mecanismos de acción. Por ejemplo la combinación de antibióticos beta lactámicos y ácido clavulánico (inhibidor de la enzima bacteriana beta lactamasa) ha sido muy eficiente. Sin embargo, han comenzado a aparecer bacterias con beta lactamasas refractarias a la inhibición por ácido clavulánico. También se puede modificar la estructura de antibióticos existentes para intentar evadir el mecanismo de resistencia, pero es difícil lograrlo sin afectar la actividad del antibiótico.
b) Identificar nuevas dianas
Una manera de desarrollar nuevas armas es identificar nuevos blancos (es decir, nuevas moléculas o vías metabólicas donde el antibiótico puede actuar). Existen unas 200 proteínas esenciales y conservadas en las bacterias, pero el número de dianas reconocidas por los antibióticos disponibles es muy bajo. Gracias a la secuenciación del genoma de numerosas bacterias y a las herramientas bioinformáticas, se pueden identificar in silico (es decir, por medio de la simulación en ordenadores) nuevas dianas contra las cuales diseñar antibióticos. Por ejemplo, comparando el genoma de Streptococcus pneumoniae con una bacteria relacionada pero de vida libre (S. thermophilus) se han identificado dos genes patógenos (expresados por la primera pero no por la segunda), candidatos para el diseño de fármacos que los inactiven.
c) Buscar nuevas armas en la naturaleza
Por el momento ningún antibiótico en el mercado viene del océano y sin embargo el mar representa la fuente más importante -e inexplorada- de fármacos naturales en el planeta. Las algas, las esponjas y los microorganismos del fondo marino son sólo algunos ejemplos de organismos a partir de los cuales se han aislado ya compuestos con actividad antimicrobiana. Por ejemplo, una bacteria recogida cerca de la costa de Santa Barbara, California (Streptomyces sp) produce anthracimicina, un compuesto capaz de matar a cepas de Staphylococcus aureus resistentes a la meticilina (SARM).
NoMorFilm, buscando antibióticos en el mar
Desde ISGlobal lideramos el proyecto europeo NoMorFilm, cuyo objetivo es encontrar, a partir de microalgas marinas, nuevas moléculas con actividad antibiótica y antibiofilm (películas bacterianas muy difíciles de tratar con antibióticos existentes).
Las bacterias que viven en el medio ambiente, sobre todo aquellas que se encuentran en el suelo o en ambientes extremos, representan igualmente una fuente rica pero poco explorada de compuestos antimicrobianos. Muchas de ellas son difíciles de cultivar en el laboratorio, pero gracias a técnicas como la transferencia de ADN y la metagenómica, se puede secuenciar su genoma e identificar candidatos con actividad antibacteriana.
Nuestro cuerpo también alberga una gran cantidad de bacterias, virus y hongos. Sólo en nuestro intestino viven trillones de bacterias (más de mil especies diferentes con más de 3 millones de genes). Esta microbiota no sólo no nos perjudica sino que frecuentemente nos protege de bacterias patógenas. Por ejemplo, se ha encontrado que el estafilococo que vive en nuestra nariz produce un péptido (lugdunina) que impide la colonización por S. aureus.
Entre las diversas opciones que ofrece la naturaleza para luchar contra las bacterias resistentes destacan la búsqueda de fármacos naturales en el mar, las bacterias que viven en el medio ambiente, nuestra propia microbiota o los bacteriófagos
La necesidad de nuevos agentes antimicrobianos ha vuelto a despertar interés en los bacteriófagos, hasta ahora estudiados y utilizados principalmente en la antigua URSS y Europa del este. Estos virus, que infectan y destruyen específicamente a bacterias, son eficientes –al menos en el laboratorio- para combatir películas bacterianas (biofilms). Los bacteriófagos también se pueden usar en combinación con otros antibióticos: por ejemplo, el fago OMKO1 se adhiere a la bomba que la bacteria Pseudomonos aeruginosa usa para expulsar los antibióticos, haciéndola más vulnerable a los mismos. El problema es que la terapia por fagos requiere saber de qué bacteria se trata. Además, la producción de lotes estables de fagos es difícil, y su eficacia para tratar infecciones humanas todavía no se ha demostrado con ensayos clínicos controlados.
A partir de diversos organismos en la naturaleza, se ha podido obtener péptidos naturales (cadenas de aminoácidos más pequeñas que una proteína) muy prometedores. Las endolisinas, unas enzimas derivadas de los bacteriófagos y que degradan la pared celular de la bacteria, se han usado con éxito en pruebas clínicas para tratar infecciones cutáneas. En ISGlobal, hemos aislado a partir de la piel de la rana seis péptidos con actividad contra bacterias resistentes a la colistina.
Se han logrado resultados muy prometedores con simples trasplantes de microbiota fecal de un individuo sano a un paciente con infecciones recurrentes por 'Clostridium difficile'
Una alternativa a matar las bacterias patógenas es “desarmarlas” para hacerlas menos agresivas. Para ello, se buscan moléculas capaces, por ejemplo, de interferir con la secreción de toxinas o la infección del huésped. Muestra de ello son los pilicidas, unos compuestos que interfieren en la formación de pili, una estructura implicada en la adhesión de E. coli a la célula huésped. Al no matar a la bacteria, las moléculas antivirulencia tienen la gran ventaja de no ejercer una presión selectiva tan grande como los antibióticos y ralentizar así la aparición de resistencias. También podrían contribuir a disminuir la cantidad de antibióticos necesarios cuando se administran en combinación con los mismos. Se trata de una táctica prometedora pero para la cual aún no hay moléculas en fase de estudios clínicos.
Finalmente, otra táctica- totalmente distinta- consiste en reemplazar unas bacterias por otras. Se han logrado resultados muy prometedores con simples transplantes de microbiota fecal de un individuo sano a un paciente con infecciones recurrentes por Clostridium difficile, una bacteria que causa diarreas graves.
2. Reconocimiento del enemigo
(diagnóstico)
¿Por qué es fundamental y qué se necesita?
Así como en una batalla es importante tener información sobre el enemigo antes de atacarle (de cuántos efectivos dispone, qué tipo de armas tiene, etc.), en la guerra contra las resistencias microbianas es fundamental saber, lo más rápidamente posible, si una infección es bacteriana o no, y a qué bacterias y resistencias nos estamos enfrentando para escoger el tratamiento adecuado. Normalmente, las técnicas tradicionales de cultivo requieren entre 48 y 72 horas para aislar e identificar el agente patógeno. Una detección en tiempo real permite adaptar inmediatamente el tratamiento a la(s) bacteria(s) en cuestión. Con esto, no sólo se mejoraría el tratamiento del paciente, sino también se reduciría el uso innecesario de antibióticos de amplio espectro, lo cual contribuye al desarrollo de resistencias.
Un diagnóstico en tiempo real permite adaptar inmediatamente el tratamiento del paciente y evitar el uso innecesario de antibióticos
Además de rápida, la técnica de diagnóstico ideal debe ser sensible (detectar la presencia de la bacteria causante de la infección pese a que haya una cantidad pequeña) y específica (no confundirla con otras bacterias semejantes); darnos información sobre la presencia y tipos de genes de resistencia; poderse hacer en países de recursos medios y bajos (sin requerir personal o equipo altamente especializado); y tener un precio asequible. Sobra decir que la técnica ideal aún no existe (gráfico 3), pero a continuación citamos algunos esfuerzos que se están haciendo para avanzar hacia ese objetivo.
¿Qué se está haciendo?
La detección de ADN bacteriano por medio de su amplificación por la técnica de PCR (reacción en cadena de la polimerasa) es una de las herramientas de diagnóstico que más ha impactado el diagnóstico microbiológico, sobre todo por su rapidez – en un laboratorio de rutina, el tiempo necesario para identificar al patógeno y detectar posibles resistencias se puede reducir a menos de dos horas.
En ISGlobal, por ejemplo, hemos puesto a punto la detección rápida de los principales genes de resistencia en bacterias gramnegativas, las beta-lactamasas de espectro extendido y las carbapenasas, directamente a partir de hemocultivos de pacientes, y en apenas 15 minutos. También hemos podido detectar, a partir de sangre extraída de un catéter, la presencia de SARM en 90 minutos.
El principal reto de las pruebas de detección por amplificación de ADN es el rango relativamente limitado de mecanismos de resistencia que se pueden detectar. Otro problema es que, incluso cuando se detecta la presencia de un gen resistente, éste puede no expresarse a niveles suficientemente elevados para traducirse en una resistencia funcional.
Una técnica emergente y muy prometedora conocida por las siglas MALDI-TOF/MS, permite identificar rápidamente diferentes especies de bacterias, e incluso diferenciar linajes dentro de una misma especie. La metodología se basa en la espectrometría de masas, que identifica el conjunto de proteínas de una bacteria en función de su masa. En ISGlobal hemos logrado desarrollar un protocolo automatizado de MALDI-TOF para clasificar cepas de Staphylococcus aureus resistentes a la meticilina en cuestión de minutos. De manera importante, es un protocolo que se puede implementar en laboratorios de rutina que ya realizan la identificación bacteriana por espectrometría de masas.
Sin embargo, para que estas técnicas puedan usarse en países de bajos y medios recursos, todavía queda camino por recorrer: es necesario desarrollar plataformas y kits comerciales que reduzcan la necesidad de personal especializado, y que sean accesibles y asequibles.
3. Tácticas del adversario
(los mecanismos de resistencia)
¿Por qué es un campo de batalla fundamental?
Entender los mecanismos por los cuales las bacterias se defienden del ataque antibiótico es esencial para desarrollar maneras de aumentar la eficacia e incrementar la vida de los antibióticos disponibles, así como minimizar la aparición de resistencias hacia nuevos compuestos.
Una bacteria puede defenderse de la “agresión” causada por un antibiótico a través de cuatro mecanismos principales:
a) Impedir su entrada disminuyendo la cantidad de canales que usan los antibióticos para penetrar en la bacteria. Una disminución en la cantidad de canales (llamados porinas) está asociado con la resistencia a las quinolonas.
b) Destruirlo o inactivarlo una vez que ha entrado, gracias a la acción de proteínas llamadas enzimas: la producción de beta lactamasas por las bacterias es uno de los principales mecanismos de resistencia hacia los beta lactámicos, antibióticos ampliamente utilizados que inhiben la síntesis de la pared celular.
c) Modificar la diana o la vía metabólica sobre la cual actúa el antibiótico: las bacterias resistentes a la vancomicina presentan una alteración en el sitio donde normalmente se une el antibiótico, impidiendo su acción.
d) Expulsarlo hacia el exterior, gracias a un sistema de bombas de flujo: este es uno de los principales mecanismos que usa la bacteria Pseudomonas aeruginosa para defenderse de los aminoglucósidos.
Las bacterias pueden adquirir genes que les confieren resistencia a antibióticos por los siguientes mecanismos:
a) Mutaciones espontáneas en su material genético (ADN)
b) Transferencia de genes de resistencia entre bacterias a través de la:
- Transformación: la bacteria captura ADN del medio ambiente
- Conjugación: es el intercambio de plásmidos (pequeños círculos de ADN) que contienen genes de resistencia y que pueden pasar de un tipo de bacteria a otra. Esta transmisión horizontal de genes es altamente preocupante, ya que un solo plásmido puede conferir varias resistencias diferentes. En 1968, 12.500 personas en Guatemala murieron de una epidemia de diarrea por Shigella. La bacteria albergaba un plásmido con genes que conferían resistencia a cuatro antibióticos. En EEUU, se detectó en una mujer con infección urinaria por E. coli, que contenía 15 genes de resistencia antimicrobiana en dos plásmidos. Uno de esos genes es mcr-1, que confiere resistencia al antibiótico de última línea colistina.
- Transducción: se transfieren genes de resistencia de una bacteria a otra a través de bacteriófagos (virus que infectan a las bacterias)
¿Qué se está haciendo?
Es necesario continuar caracterizando los mecanismos y las consecuencias de la resistencia antimicrobiana, sobre todo aquellos usados por las bacterias que más problemas causan en el contexto hospitalario. Por ejemplo, en ISGlobal hemos encontrado que cuando el patógeno nosocomial Acinetobacter nosocomialis adquiere resistencia a la colistina, sufre cambios en la composición de su membrana externa que le hacen perder en virulencia, de la misma manera que la resistencia a las quinolonas en Salmonella enterica reduce la expresión de genes asociados a la virulencia y disminuye su capacidad de infección. Estos resultados, aunque alentadores, no se pueden generalizar.
Es fundamental entender el camino que lleva a la adquisición de resistencias, pero también prever futuros mecanismos de resistencia
Entender el camino que lleva a la adquisición de resistencias es otro aspecto fundamental. Por ejemplo, hemos analizado la evolución de resistencias de Pseudomonas aeruginosa aisladas de pacientes en cuidados intensivos, demostrando que se necesita la aparición gradual de varios mecanismos para llegar a un “punto de quiebra” más allá del cual se pierde sensibilidad a los antibióticos. Cuando Streptococcus pneumoniae es sometida a dosis crecientes de antibiótico, la respuesta inmediata requiere mutaciones en una pequeña cantidad de genes (menos de 10), pero el problema es que dos cepas pueden llegar al mismo resultado (supervivencia y crecimiento) usando diferentes genes.
Por eso también es importante tratar de ir un paso más allá del adversario y prever futuros mecanismos de resistencia. Cada vez hay más evidencia de que los genes de resistencia a antibióticos están ampliamente distribuidos y que muchos ecosistemas asociados con la agricultura como el suelo, las aguas residuales, o el estiércol son reservorios naturales de resistencias potenciales. Estudios de metagenómica a partir de estos reservorios comienzan a aportar valiosa información sobre la posible aparición de nuevas resistencias. Por ejemplo, el análisis de Staphylococcus aureus resistentes a la meticilina aislados a partir de playas recreativas o superficies tocadas frecuentemente por personas, muestra que 98% de ellos son resistentes a otros antibióticos además de la meticilina, reflejando la “reserva” de genes de resistencia en el ambiente.
4. Los servicios secretos
(vigilancia)
¿Por qué es un campo de batalla fundamental?
Se suele decir que, a la larga, una guerra se gana gracias a los servicios de información e inteligencia. Lo mismo aplica para la guerra contra la resistencia bacteriana. Los estudios de vigilancia proporcionan valiosa información para detectar tendencias en el tipo y la frecuencia de microorganismos causantes de infecciones en humanos y en los mecanismos de resistencia antimicrobiana, a nivel nacional y global, y desarrollar intervenciones específicas y adaptadas a cada contexto. Además, la comparación de datos a lo largo del tiempo permite evaluar la eficacia de dichas intervenciones.
¿Qué se está haciendo?
La vigilancia consiste en monitorear y detectar la aparición de nuevas cepas resistentes. En este sentido, desde ISGlobal hemos logrado reconstruir la diseminación de un gen de resistencia a los carbapenemos (OXA-23) en la bacteria A. baumannii, en diferentes países de la cuenca Mediterránea a lo largo del tiempo. Así mismo, hemos caracterizado una de las primeras cepas de A. pittii portadora del gen NDM-1 (una beta lactamasa descrita primero en Nueva Delhi y que confiere resistencia a los carbapenemos), aislada de una paciente en Turquía. Los resultados del estudio sugieren que el gen NDM circuló en otras especies de Acinetobacter, antes de propagarse a especies de relevancia médica.
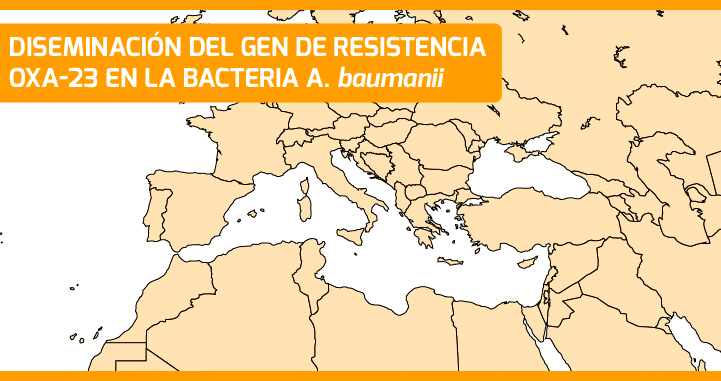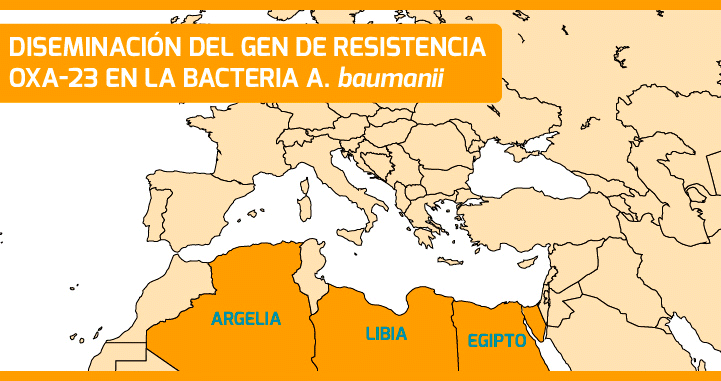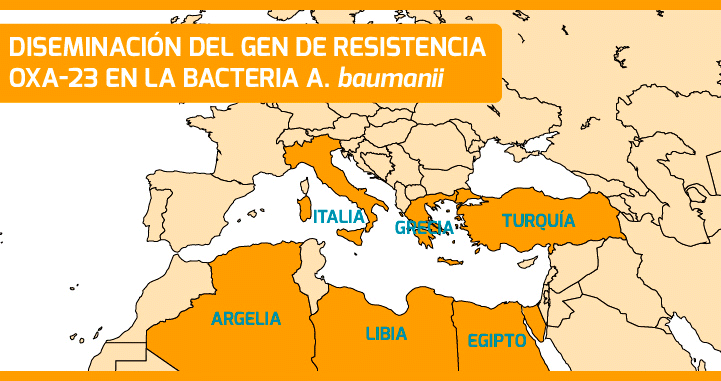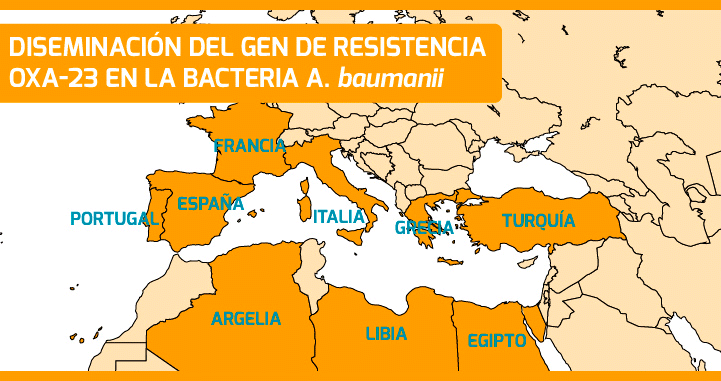
La vigilancia de resistencia antimicrobiana a nivel global necesita una mayor coordinación y armonización
Estudios como este, provenientes de múltiples hospitales y centros de salud, sirven para alimentar redes y proyectos a nivel nacional y regional cuya misión es colectar datos sobre la prevalencia de resistencias. Por ejemplo, en Europa existe la EARS-Net, una red de sistemas de vigilancia nacionales que reúne datos de 900 laboratorios que dan servicio a más de 1400 hospitales (y a unos 100 millones de ciudadanos europeos). La red recoge datos sobre susceptibilidad antimicrobiana de las siete bacterias que causan el mayor número de infecciones en humanos (Streptococcus pneumonia, Staphylococcus aureus, Enterococcus faecalis, Enterococcus faecium, Escherichia coli, Klebsiella pneumonia, Pseudomonas auruginosa).
Muchas de estas redes también proporcionan datos sobre las tendencias en tipos y dosis de antibióticos prescritos, lo cual permite relacionar el uso de ciertos antibióticos con el aumento o disminución de ciertas resistencias y diseñar protocolos que ralenticen la aparición de las mismas.
Sin embargo, como señalado por la OMS en su último reporte, la vigilancia de resistencia antimicrobiana a nivel global necesita una mayor coordinación y armonización, y aún quedan muchas lagunas de conocimiento en torno a bacterias importantes a nivel de salud pública (por ejemplo, la tuberculosis multirresistente).
Conclusión
La aparición de genes que pueden conferir resistencia a diferentes antibióticos es algo que no podemos evitar – forma parte del proceso evolutivo normal de todo microorganismo. Sin embargo, lo que sí podemos evitar es seleccionar a las bacterias que expresan dichos genes como resultado del abuso y mal uso de los antibióticos, actuales y futuros.
La buena noticia es que tenemos conocimientos, armas y estrategias prometedoras para vencer, o por lo menos controlar, la aparición y propagación de bacterias multirresistentes en un futuro cercano. Las bacterias tienen un gran número de dianas (moléculas y vías metabólicas) potenciales que aún no explotamos. El mar y otros recursos naturales nos ofrecen una fuente increíblemente diversa de productos con potencial antibacteriano, resultado de millones de años de selección natural. El desarrollo de nuevas técnicas de diagnóstico nos permite adaptar cada vez más el tratamiento a cada paciente y evitar el uso innecesario de antibióticos. Estamos comenzando a conocer los mecanismos que conducen a la aparición y selección de resistencias, y sus consecuencias. Y, por último pero no por ello menos importante, estamos construyendo y fortaleciendo redes de vigilancia de propagación y aparición de resistencias antimicrobianas.
La investigación científica, pues, es un componente esencial en la guerra contra la resistencia antimicrobiana. Sin embargo, no es el único. También se requiere la sensibilización de la población y la aplicación de leyes que garanticen el uso racional de los antibióticos (incluida la agropecuaria) e incentiven el desarrollo de futuros fármacos. Sin ello, por muchos soldados y armas que tengamos, no ganaremos la batalla.












